Der Betrug mit dem PCR-Test
Über einen Test, mit dem man eine Pandemie herbeizauberte.
Wenn man heutzutage in den Medien von Coronatests spricht, ist im Allgemeinen der sogenannte PCR-Test gemeint. Derzeit wird auch an der Einführung von Antikörper- und Antigentests gearbeitet, aber diese Verfahren waren bis weit in den Herbst 2020 hinein eindeutig in der Minderzahl. Erstaunlicherweise weiß das Gros der Bevölkerung nicht, was der PCR-Test tatsächlich tut — und was er nicht tut. Das ist fatal, denn was er NICHT tut, ist das, was ihm in Politik und Medien fortlaufend angedichtet wird. Der gefeierte PCR-Test des Christian Drosten weist keine Infektionen und auch nicht das “neuartige Virus” nach.
Wenn gleich das geschickterweise nicht so direkt gesagt wird, so ist doch die Berichterstattung der Meinungsführer ganz klar darauf ausgerichtet, die PCR-Methode als eine zu verstehen, welche Infektionen — unter anderem mit dem “neuartigen Virus” — nachweisen könnte. Unverblümt wird Tag für Tag, Stunde für Stunde die Mär von den “Neuinfektionen” verbreitet. Auf diese Art und Weise verbreitet man Angst und Hysterie, um als Preis zur vermeintlichen schnellen Rettung vor dem Virus eine Fülle politischer Entscheidungen zu akzeptieren, die noch vor Monaten undenkbar, ja schlicht nicht umsetzbar gewesen wären.
Es muss bei der Betrachtung dessen, für was die PCR-Methode taugt und für was sie das nicht tut — so auch wie deren Ergebnisse für wirtschaftliche, politische und letztlich ideologische Zwecke benutzt werden — nicht die Frage nach der Existenz des “neuartigen Virus” gestellt werden. Wenn wir über die grundlegende Funktion des Tests Bescheid wissen, wird uns auch klar, warum dem so ist.
Das ist von fundamentaler Bedeutung für alles, was um die im März des Jahres 2020 ausgerufene Pandemie geschehen ist. Denn wenn wir genau hinschauen, ist allein der PCR-Test die Grundlage, auf dem die “Coronavirus-Pandemie” aufbaut. Dabei sind nicht die Ergebnisse des Tests das Problem — der Test funktioniert sehr wohl zuverlässig —, sondern die Art und Weise ihrer Herbeiführung wie Interpretation.
Vom SARS-CoV-2 — Virus
Der Steckbrief zum “neuartigen Virus” (a1) wurde am 29. Januar 2020 von chinesischen Forschern im Wissenschaftsportal Lancet veröffentlicht. Grundlage waren die Proben von neun an den Atemwegen erkrankten Chinesen. Acht davon hatten zuvor den inzwischen allbekannten Meeresfrüchtemarkt von Huanan in der chinesischen Metropole Wuhan besucht (1).
Was dem Autor beim Studium des Lancet-Berichts auffiel, ist die Tatsache, dass zwar von Isolaten gesprochen wird, von der vollständigen Isolierung eines Virus jedoch keine Rede sein kann. Dies widerspricht allerdings dem Kochschen Postulat für den zweifelsfreien Nachweis eines Erregers, samt seiner vorgeblich krankmachenden Eigenschaften — dazu gleich noch mehr.
Auch wurde dieser Bericht publiziert, NACHDEM der an der Berliner Charité wirkende Christian Drosten “seinen” PCR-Test für das “neuartige Virus” bereits am 16. Januar veröffentlicht hatte. Es ist keinesfalls transparent, woher das Team von Drosten im Vorab die Daten für das Virus bezog, um daraus in Rekordgeschwindigkeit einen Test zu kreieren. Proben mit einem vollständigen Virus-Isolat standen ihm jedenfalls nicht zur Verfügung (2).
Die Herangehensweise zur angeblichen Entdeckung des SARS-CoV-2 — Virus war vom Grundsatz her die Gleiche, wie sie jetzt beim “Nachweis des Virus” mittels PCR-Test zu sehen ist.
In einem ersten Schritt züchteten die chinesischen Wissenschaftler sogenannte Isolate aus den Proben der Patienten (3). Dieses Züchten ist ein Thema für sich, denn so klar ist es nicht, was die chemischen und biochemischen Substanzen (unter anderem Antibiotika), die man in die Proben einbringt, tatsächlich bewirken.
Mittels sogenannter Transkription und Amplifizierung der Isolate (a1) wurde das aus dem “Isolat” entnommene Erbmaterial durch Teilung vermehrt. Transkription beschreibt die Überführung der einstrangigen und somit nicht eigenständig reproduktionsfähigen RNS (Ribonukleinsäure) in die Aufspaltung einer doppelstrangigen DNS (Desoxyribonukleinsäure), jener DNS, die als Erbinformation in unseren Zellkernen abgelegt ist (4). Amplifikation meint die zyklisch vorgenommene Vermehrung (Kopieren) von RNS mittels Einbindung in die DNS (5, a2).
Von einer auch nur annähernd den Kochschen Postulaten gerecht werdenden vollständigen Isolation des “neuartigen Virus” kann jedoch keine Rede sein. Die chinesischen Wissenschaftler schrieben in ihrer Veröffentlichung (alle Hervorhebungen in den Zitaten durch Autor, a3):
“Von diesen [neun] Personen erhielten wir vollständige und teilweise 2019-nCoV-Genomsequenzen. Die Viruskontigstellen wurden mittels Sanger-Sequenzierung miteinander verbunden, um die Genome in voller Länge zu erhalten, wobei die terminalen Regionen durch schnelle Amplifikation der cDNA-Enden bestimmt wurden.” (1i)
Die Sanger-Sequenzierung ist eine technische Methode zur gerade erwähnten Aufspaltung von DNS, um an einen der nun “offenen” DNS-Stränge bestimmte RNS koppeln zu können (6). Die Sequenzierung von RNS-Sequenzen ist nicht das Gleiche wie die Sequenzierung eines vollständig isolierten Virus. Und tatsächlich findet der Forschende reichlich Informationen zur Sequenzierung in Bezug auf SARS-CoV-2, nie aber auf das Virus als Ganzes (7):
“Die zehn Genomsequenzen von 2019-nCoV, die von den neun Patienten erhalten wurden, waren äußerst ähnlich und wiesen eine Sequenzidentität von mehr als 99-98% auf.” (1ii)
Wie hat man dann aber das Virus entdeckt, so doch eine vollständige Isolierung zwar behauptet, dies aber nirgends nach wissenschaftlichen Kriterien dokumentiert ist?
Man hat die in den Patienten gefundenen Sequenzen um fehlende “Bausteine” ergänzt und zusammengefügt. SARS-CoV-2 wurde — wie andere Viren auch (8) — unter Verwendung komplexer mathematischer Algorithmen in Computer-Software modelliert. Es wurde aus vollständigen Gensequenzen wie auch Genfragmenten konstruiert, nicht einmal rekonstruiert — aber nicht nur das. Denn tatsächlich hat man offensichtlich sogar “passenden”, genetischen Code “erfunden” (oder in Datenbanken “gefunden”), um ein vollständiges Modell des Virus zu erhalten. Die Virologen nennen dieses Software-gestützte Verfahren Alignment (9) und behaupten, dass sich die Ausrichtung/Zuordnung — so die deutsche Entsprechung von Alignment — an eine Vorlage in Form eines real existierenden, vollständig isolierten Virus hält.
“Sequence alignment of 2019-nCoV with reference sequences was done with Mafft software (version 7.450).” (10)
Es sollte klar sein, dass die Konstruktion, das Modell eines Virus etwas fundamental anderes beschreibt, als dessen Erkennung als Ganzes, als dessen jederzeit reproduzierbares Isolat. Das Virus, so es dieses gibt, ist groß genug, um jederzeit und ohne weiteres aus menschlichen Abstrichen sichtbar gemacht zu werden. Davon ist dem Autor bis heute nichts bekannt. Vereinfacht stellt sich die Konstruktion des Virus wie im folgenden Bild zu sehen dar (b1):
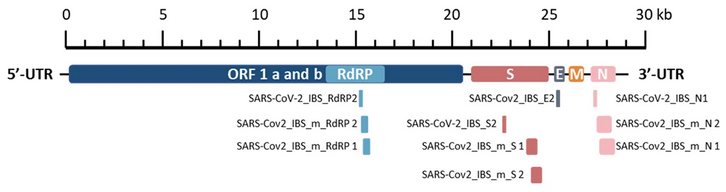
30 kb steht für 30.000 Basen (Nukleotide). Laut dem Modell gehört das SARS-CoV-2 — Virus, als Mitglied der Familie der Coronaviren mit seinen fast 30.000 aneinandergereihten Nukleotiden zu den größten (längsten) Viren. Da es in RNS gebunden ist, besteht es aus nur einem, gewundenen Einzelstrang (Helix), im Gegensatz zur Doppelhelix bei DNS (11).
Was wir hier erkennen, sind — neben nicht weiter spezifizierter RNS — mehrere, dem SARS-CoV-2 — Virus als charakteristisch zugeschriebene, spezielle Proteine: das RdRP-Gen, das S-, M-, N- und das E-Gen. Dieses (obige) Bild werden wir uns im Verlauf dieser Abhandlung nochmals zu Gemüte führen. Weil es sehr schön auf den täglichen, massenmedialen Betrug hinweist, nachdem mit der PCR-Methode angeblich auf ein Virus getestet würde — was es nicht tut (b1i).
Bedenken wir: Nicht nur ist die obige Abbildung modellhaft, sondern sie bezieht sich auch auf ein Modell, eine Abstraktion, eine Annahme, aber keinesfalls auf eine als Ganzes, als vollständiges Isolat mit der dort nachgewiesenen Struktur des Virus. Auch wenn renommierte Fachzeitschriften wie The Lancet in diversen Publikationen den Eindruck erwecken, dass nachweislich SARS-Viren isoliert worden wären, ist zu sagen: Dem ist nicht so, auch nicht beim SARS-Virus, das als Vorlage für die Modellierung des “neuartigen Virus” genutzt wurde (12, 13).
Als chinesische Forscher am 24. Januar 2020 ihre Untersuchungsergebnisse zu Patienten, die vorgeblich am SARS-CoV-2 — Virus erkrankt waren, veröffentlichten, waren sie ehrlich genug, darauf hinzuweisen:
“Da wir keine Tests zum Nachweis infektiöser Viren im Blut durchgeführt haben, haben wir den Begriff Virämie vermieden und stattdessen RNAämie verwendet. RNAämie wurde als ein positives Ergebnis für die Real-Time RT-PCR in der Plasmaprobe definiert.” (14)
Indirekt haben die Forscher damit auch klar gemacht, dass ein PCR-Test keine isolierten Viren nachweist. Tatsächlich und nachgewiesenermaßen real sind freilich die Gensequenzen, welche man dem Virus zuspricht (a3), und auf eben solche zielt auch die PCR-Methode, mit der wir uns nun befassen.
Der PCR-Test
Das PCR-Testverfahren beschreibt im Grunde eine ganze Reihe sich im Detail unterscheidender Technologien, welche aber alle als zentrale Methode den natürlichen Prozess der Polymerase-Kettenreaktion (aus dem Englischen: Polymerase Chain Reaction, daher kurz PCR) beinhalten. Für den Nachweis bestimmter Gensequenzen, die dem SARS-CoV-2 – Virus zugeschrieben werden, kommt die RT-PCR – Methode zur Anwendung. Das hat insbesondere damit zu tun, dass es sich beim zu detektierenden Material nicht um DNS sondern RNS handelt. Die Methode gliedert sich grundsätzlich in drei Prozesse:
- Extraktion (Reinigung, “Isolation”) des Materials aus der Probe
- Transkription (Überführung, Umschreiben, Enzymierung) der RNS der Gensequenz in cDNS
- Amplifizierung (Vermehrung) der cDNS/DNS (15, 16)
Was geschieht dabei nun im Einzelnen?
Extraktion
Man sollte annehmen, dass ein Erreger aus einer Patientenprobe (Blut, Speichel, Gewebepartikel) mittels geeigneter Verfahren direkt isoliert wird, um ihn nachzuweisen. Isoliert meint hier, dass er als mikrobiologisches Objekt vollständig und eindeutig abgrenzbar von seinem Milieu und umgebenden wie interagierenden Mikroben erfasst wurde. Davon kann bei der vermeintlichen Extraktion des SARS-CoV-2 – Virus — die irritierender Weise aber auch als Isolation bezeichnet wird — keine Rede sein.
Die sogenannte Isolation des “neuartigen Virus” geschieht außerhalb des menschlichen Körpers, in dem die Probe in vitro (in einem Reagens) auf aggressive Art und Weise chemisch und biochemisch bearbeitet wird. Dieser Vorgang wird auch als Reinigung bezeichnet. Die im Ergebnis diesem Cocktail entnommene Substanz bringt aber nun eben auch keine (isolierten) Viren ans Licht — und das behaupten die Virologen interessanterweise auch nicht. Das Extrakt gewinnt man:
“[d]urch Vergiftung mit zelltoxischen Antibiotika, durch extremes Verhungern mittels Entzug der Nährlösung und durch Zugabe von verwesenden, also sich zersetzenden und dabei giftigen Stoffwechselprodukte freisetzenden Eiweißen.” (17)
Den Proben setzt man Substanzen wie zum Beispiel Triphosphat, Magnesiumsulfat, Isopropanol, Kohlendioxid, Chloroform, Serum aus Rinderföten und desweiteren Antibiotika wie Penicillin und Streptomyzin zu, während diese wiederholt zentrifugiert und mit starken Temperaturschwankungen bis hin zum Gefrierpunkt gestresst werden (18 bis 20). Wie bereits weiter oben erwähnt, ist dieses “Isolieren” adäquat zum Verfahren, dass die chinesischen Wissenschaftler bei ihrer “Entdeckung” des Virus benutzten:
“Proben der bronchialen Lungenflüssigkeit wurden in sterilen Behältern gesammelt, denen Virustransportmedium [chemische — und biochemische Substanzen, Antibiotika] hinzugefügt wurde. Die Proben wurden dann zentrifugiert, um Zelltrümmer zu entfernen. Der Überstand wurde auf humane Epithelzellen der Atemwege beimpft, die aus Atemwegsproben von Patienten gewonnen wurden, die sich einer Operation wegen Lungenkrebs unterzogen hatten […].” (3i)
Da wird also eine organische Substanz extrem gestresst, abgetötet und zertrümmert — und dann bezeichnet man das vom Reagensboden entnommene Refugium als Isolat? Woher um alles in der Welt nimmt man die Gewissheit, dass da Viren drin sind und dass diese die Tortur überlebt hätten? Ersetzt hier Gewissheit echtes Wissen?
Wenn das, was bei der Extraktion geschieht, die Isolation von Viren ermöglichte: Wozu benötigt man dann noch die weiter unten beschriebene Polymerase-Kettenreaktion? Der Laie meint: Die Elektronenmikroskopie müsste in der Lage sein, in einem solchen Extrakt Viren zweifelsfrei — also ohne die Gefahr einer Verwechslung mit anderen Mikroben — aufzunehmen. Das ist ganz offensichtlich nicht der Fall. Die Extraktion liefert einen Friedhof zertrümmerter Mikroben — und diese Trümmer sind natürlich nachweisbar, nämlich als relativ kurze Fragmente von DNS und RNS.
Es ist nämlich so, dass die PCR-Methode mittels der Sanger-Sequenzierung bis zu 3.000 Nukleotide lange Gensequenzen nachzuweisen in der Lage ist (21). Durch Einsatz spezieller Polymerasen-Gemische, sowie weiterer Zusätze in der PCR lassen sich sogar Sequenzen von über 20.000 Basenpaaren vervielfältigen (15i).
Jedoch: Auch wenn die Methode extrem sensitiv arbeitet und fast aus dem Nichts Gene vervielfältigen (amplifizieren) kann, so ist doch das “fast” wichtig. Eine Winzigkeit vom Gesuchten muss schon vorliegen. Dass allerdings aus einem wie in oben beschriebener Art und Weise gewonnenen Extrakt ein komplettes, aktives Gen nunmehr per PCR sequenziert und “isoliert” werden kann, ist für den Autor mehr als fraglich.
Aber Fragmente lassen sich sehr wohl transkribieren und amplifizieren (überführen und vermehren, siehe weiter unten). Fragmente, Trümmer von Mikroben, sind — wie das bei Trümmern nun einmal so ist — sehr klein. In RNS beziehungsweise DNS übersetzt heißt es, dass die Gensequenzen sehr, sehr kurz sind.
Je kürzer die gesuchte Gensequenz und je höher die Anzahl der Zyklen bei der Amplifizierung (Vermehrung), desto größer ist auch die Wahrscheinlichkeit, fündig zu werden. Und darum ging es auch, als der PCR-Test für medizinische Zwecke entdeckt wurde: nämlich etwas Bekanntes auf jeden Fall zu finden (a4). Je kürzer und simpler jedoch die Struktur, desto höher ist auch die Chance, sie rein nach dem Zufallsprinzip aufzuspüren. Einfach deshalb, weil diese mit abnehmender Länge immer beliebiger wird.
Aber auf die knapp 30.000 Nukleotide, die man SARS-CoV-2 zuschreibt (22, 23), ist der Test von seiner gesamten Konzeption her überhaupt nicht angelegt. Schon daher ist es mindestens eine hochgradig schlampige Aussage, dass der Test Viren erkennen würde.
Dass der Test statt eines vollständigen Genoms nur Teile dessen, RNS-Sequenzen sucht, ist es aber nicht allein. Denn die RNS, die er sucht, ist — entsprechend seinem Ansatz, sicher fündig auf etwas Vermutetes zu werden — auch noch extrem kurz. Wie die RNS aufgespürt wird, wollen wir uns im Weiteren anschauen.
Reverse Transkription
Das im Zuge von SARS-CoV-2 angewandte Verfahren koppelt die PCR (Polymerase-Kettenreaktion) mit einem zweiten Verfahren, das — so wie das erste — natürlichen Prozessen der Vermehrung von Erbsubstanz entlehnt ist. Damit sprechen wir von einer sogenannten RT-qPCR (a5), bei der das Enzym Transkriptease eine Schlüsselrolle spielt. Die reverse Transkription (Reverse-Transkriptase oder RT) ist notwendig, weil die einstrangige RNS des gesuchten Gens (oder einer Sequenz desselben) sich nicht teilen kann, so wie eine Doppelstrang-DNS. Die RNS kann aber an DNS-Stränge “andocken”, wenn wiederum diese geteilt werden (b2):
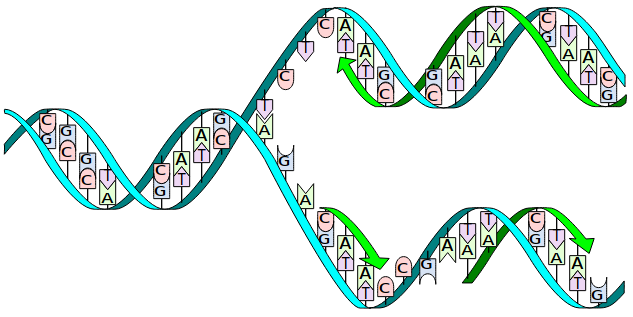
Grundbausteine der DNS sind Nukleotide, bestehend aus Phosphatresten, einem Zucker, der Desoxyribose (beide in der Abbildung nicht enthalten), sowie Paaren von jeweils zweien von vier Nukleinbasen. Was wir in Abbildung 2 sehen, ist die Doppelstrang-Helix einer DNS, die sich geteilt hat und an der nun wieder freie Nukleinbasen anlagern, zum Beispiel auch aus RNS. Die vier Nukleinbasen können sein: Adenin (A), Thymin (T), Guanin (G) und Cytosin (C). Die Kombination dieser Basen bildet die genetische Information des Trägers ab.
Die biochemischen Charakteristika der Nukleinbasen lassen im Allgemeinen nur bestimmte Anbindungen freier Basen an einen “geöffneten” DNS-Strang zu. So verbindet sich Adenin stets mit Thymin und Guanin mit Cytosin. An welchen speziellen Abschnitten diese Anbindungen ihren Anfang nehmen, dafür sorgen die Primer, sehr kurze, charakteristische Nukleotide (24).
Wenn man also Gensequenzen mittels PCR und RT finden möchte, ist es notwendig, dass die DNS ein ganz bestimmtes Design aufweist, das komplementär zur gesuchten RNA ist. Das heißt, dass sich die DNS-Stränge durch eine ganz bestimmte Abfolge von Basenpaaren auszeichnen müssen. Basenpaare, die so angeordnet sind, dass RNS eben auch an diese anbinden kann. Es ist wie mit einem Schlüssel, der halt nur in das “richtige” Schloss passt.
Der als Goldstandard gepriesene PCR-Test aus der Berliner Charité schaut im Suchtest nach dem E-Gen, genauer nach der 76 Basen langen E2-Gensequenz (25, a6), welche folgende Abfolge von Basen aufweist:
- ACAGGTACGTTAATAGTTAATAGCGT
- ACACTAGCCATCCTTACTGCGCTTCG
- ATTGTGTGCGTACTGCTGCAATAT (26)
Um an “passende” DNS-Stränge anbinden zu können, sorgt das Enzym der Transkriptease zuvor für ein Umschreiben. Eine der vier Nukleinbasen von RNS, Uracil (U), das in DNS nicht vorkommt, wird dabei durch die Transkriptease in Thymin (T) umgewandelt.
Damit kann die so umgeschriebene Sequenz nun an eine komplementäre Sequenz einer aufgespaltenen DNS anbinden. Weil Adenin stets mit Thymin und Guanin mit Cytosin bindet, stellt sich der Autor den DNS-Komplementärstrang, an dem die zuvor umgeschriebene RNS andocken kann, also folgendermaßen vor:
- TGTCCATGCAATTATCAATTATCGCA
- TGTGATCGGTGGAATGACGCGAAGC
- TAACACACGCATGACGACTTATA
Deshalb nennt man die so gebildete, neue Doppelhelix auch cDNA (complementary DNA), also komplementäre DNS (27). Mittels sogenannter Ribonukleasen und DNS-Polymerase — die hier nicht weiter behandelt werden — erfolgt danach die Synthese des zum gerade gebildeten cDNA-Stranges komplementären DNS-Stranges (28). An dieser Stelle sei nun eine weiter oben bereits angezeigte Grafik nochmals präsentiert (b1ii):

Was fällt auf?
Die Sequenz SARS-Cov2_IBS_E2 ist so ziemlich die Kürzeste, im gesamten Konstrukt des SARS-CoV-2 – Virus. Sie gerät aufgrund eben dieser Kürze in die Nähe von Beliebigkeit und eine exklusive Zuordnung zum Virus — so es ihn tatsächlich gibt — darf schwer in Frage gestellt werden. Ein Genfragment dieser Kürze wird aber seit Monaten als angeblicher Infektions-, ja gar als Erkrankungsnachweis mit COVID-19 deklariert.
Das ist einfach unglaublich. Nochmals: 76 “passende” Basenpaare im Ergebnis der Amplifikation (siehe weiter unten) sind ausreichend genug, um Gesunde zu Kranken zu stempeln, Menschen ihrer Freiheitsrechte zu berauben und ein ganzes Land in Panik zu versetzen.
Die Extraktion des Materials, welches für die RT-PCR verwendet wird, ist schon fragwürdig genug. Dass es sich nun auch noch um eine Minisequenz handelt, die als Pandemienachweis verwendet wird, setzt dem noch eins drauf. Das sehr kurze E-Gen wird als Charakteristikum allen Coronaviren zugeschrieben.
Damit ist dieser Suchtest mit der PCR-Methode hoch sensitiv und muss zwingend über mindestens einen, besser zwei oder drei Spezifitätstests abgesichert werden. Diese Tests weisen dann weitere, dem Virus zugeschriebene Gene nach — oder auch nicht. Trifft letzteres zu, lässt sich die Annahme, dass Teile des Virus im Material nachweisbar wären, nicht mehr aufrecht erhalten. Und genau das wird in Deutschland offenbar flächendeckend unterlassen.
Gern weist man in der (noch) meinungsbildenden Politik- und Medienszene darauf hin, dass China das Virus erfolgreich bekämpfen konnte, weil es so drakonische Maßnahmen im Rahmen der nichtpharmazeutischen Intervention durchgesetzt hätte. Aber könnte es möglicherweise auch sein, dass in diesem Land konsequent mehrere Spezifitätstests auf andere Corona-Gensequenzen gefahren werden und allein dadurch eine besondere epidemiologische Situation — die es eh nur in Wuhan zu beobachten gab — beendet beziehungsweise vermieden wurde? Die Frage muss offen bleiben.
Sensitivität und Spezifität
Womit wir zur nächsten Ungereimtheit bei der Anwendung des PCR-Tests in Bezug auf das “neuartige Virus” kommen. Erinnern wir uns dafür an die vorher erläuterte reverse Transkriptease (reverse Transkription), das Umschreiben und Anbauen der RNS-Sequenz auf einen freien DNS-Strang. Der Drosten-Test sieht im Grunde vor, dass Spezifitätstests durchzuführen sind, und zwar auf das RdRP-Gen. Auch dieses Gen ist sehr kurz, bestehend aus gerade einmal 100 Basen, und das — es sei wiederholt — bei einem Virus, das 30.000 Basen lang sein soll. Doch gilt es, einen weiteren, wichtigen Aspekt zu beachten.
Es spielt nämlich auch eine Rolle, wie hoch die Konzentration der Lösung ist, in welcher sich die Polymerase-Kettenreaktion abspielt beziehungsweise abspielen soll. Es dürfte einleuchten, dass, wenn ich einer extrem kurzen Gensequenz extrem viele Komplementärstränge zum Anbinden, anbiete, dass sich dann die Wahrscheinlichkeit deutlich erhöht, dass dies dann auch tatsächlich geschieht. Ein Primer, welcher den Start- und Endpunkt für den Prozess des Umschreibens von RNS auf einen DNS-Strang definiert, ist nochmal erheblich kürzer, als die umzuschreibende Gensequenz selbst, nämlich gerade 15 bis 25 Basen lang.
Was den von der WHO als Goldstandard, ja gar als “diagnostischen Leitfaden” (29) ausgerufenen Drosten-PCR-Test betrifft, fällt auf: Ausgerechnet beim Primer für das RdRP-Gen, mittels dem der Suchtest auf das E2-Gen falsifiziert werden soll, ist die Primerkonzentration auffallend hoch. Wenn man im Netz zu diversen PCR-Verfahren recherchiert, findet man heraus, dass bestimmte Primer-Konzentrationen in der Reaktionslösung eingehalten werden, und zwar zwischen 100 und 400 nM (nmol/l). Beim Test aus dem Hause der Charité wird dieser Wert deutlich überschritten — und zwar nicht nur für das RdRP- sondern auch für das N-Gen, das ebenfalls für Kontrolltests genutzt wird (Zeile 1 und 4 im ersten Abschnitt “RdRP gene”, a7, b3):
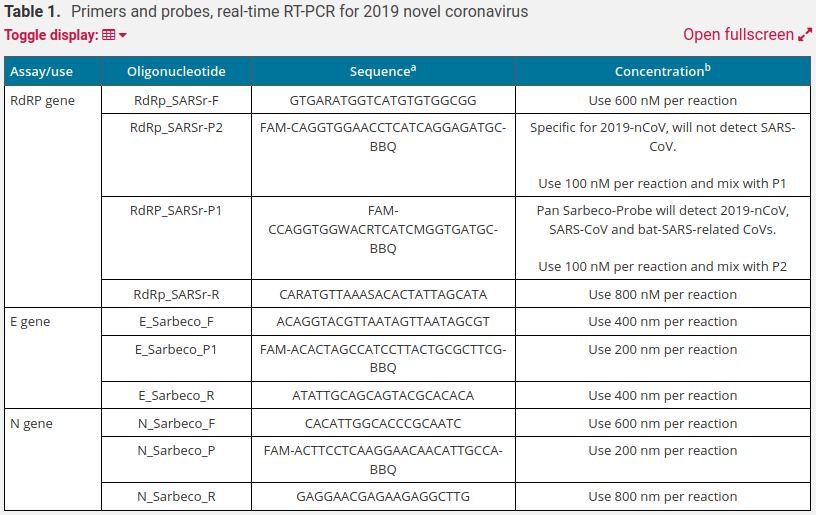
Noch etwas findet der aufmerksame Leser, wenn er sich das in der Tabelle definierte Primer-Design anschaut. Da an beide Stränge der aufgespaltenen DNS angebunden wird, gibt es also zwei Primer, einen Forward Primer für den ersten DNS-Strang und einen Reverse Primer für den Zweiten (30, Hervorhebungen durch Autor):
- GTGARATGGTCATGTGTGGCGG (RdRp_SARSr_F)
- CARATGTTAAASACACTATTAGCATA (RdRp_SARSr_R)
Wir wissen, dass genetische Informationen in den Nukleinbasen Adenin (A), Thymin (T), Guanin (G), Cytosin (C) und Uracil (U) kodiert werden. Uracil wird bei Transkription der RNS in Thymin umgeschrieben. Die obigen Primer enthalten aber jeweils einen Ausdruck ‘R’. Was bedeutet dieser?
In einer Fußnote steht dazu lapidar: “R is G/A”. Was so viel bedeutet wie: R ist entweder Guanin oder Adenin. Beide Primer können also jeweils in zwei verschiedenen Varianten an DNS binden und das, obwohl Primer bereits extrem kurz sind (19i, 31). Man nennt so etwas degenerierte Primer und tatsächlich ist es so, dass man in diesem Falle nicht weiß, wie der Primer — falls es ihn natürlicherweise gibt — wirklich aussieht (30i). Also erschafft man diese künstlich, modelliert sie am Computer (31). Damit kann so ein Primer auch an Mutationen anbinden, was die Gefahr verfälschter Ergebnisse einschließt.
Einschub: Unter anderem auf solche Ungereimtheiten sind eine ganze Reihe von Wissenschaftlern aufmerksam geworden und fordern daher von Eurosurveillance — dem Herausgeber der Publikation — die Rücknahme des Drosten-Papieres als dem eines den Prüfkriterien für wissenschaftliche Arbeiten nicht erfolgreich bestandenen Dokumentes (32 bis 35).
Interessanterweise liefern andere Staaten Primer für das RdRP-Gen, die ohne die oben beschriebenen “Platzhalter” auskommen, China zum Beispiel (36). Doch gehen wir weiter: Die Testprobe muss thermisch behandelt werden, bevor der Primer an den DNS-Strang ankoppeln kann.
Um die Teilung der DNS anzustoßen beziehungsweise zu beschleunigen, wird als Katalysator Wärme eingesetzt. Dabei werden die Proben in sogenannten Thermocyclern auf über 90 Grad Celsius erhitzt, was zur Aufspaltung (Denaturierung auch Schmelzen, im Englischen melting bezeichnet) der DNS führt. Im nachfolgenden Abkühlungsvorgang auf etwa 55 bis 65 °C (Renaturierung) können nun Startersequenzen, die Primer (Primerhybridisierung oder primer annealing) an den DNS-Strang anbinden.
Wählt man allerdings die Temperatur für diesen Prozess zu nah an oder gar unter den minimalen Grenzwert von 55 °C, dann kann das zu Fehlern, zu Mutationen führen. Primer könnten sich an eben nicht vollständig komplementäre Sequenzen auf dem DNS-Strang anlagern (37). Es ist zumindest eine Anmerkung wert, dass der PCR-Test aus dem Drosten-Team sich genau an diesem Grenzwert bewegt:
“Thermal cycling was performed at 55 °C for 10 min for reverse transcription, followed by 95 °C for 3 min and then 45 cycles of 95 °C for 15 s, 58 °C for 30 s.” (19iii)
Die Kombination degenerierter Primer (siehe weiter oben) mit solchen Grenzwerten birgt die Gefahr, dass der Test fehlerhaft arbeitet — so die Ansicht des Autors.
Auf jeden Fall binden nach den Primern nunmehr die “passenden” RNS-Sequenzen an die offen liegenden DNS-Stränge (38, 39).
Amplifizierung und Ct-Wert
Im folgenden wird ein Prozess gefahren, der die gewonnene cDNA — die komplementäre, mit den Primern und der transkribierten RNA “bestückten” DNS — zu vervielfältigen versucht. Ein aussagefähiges Ergebnis hängt unter anderem von zwei Parametern ab:
- der Menge von cDNA (komplementärer DNS), die mittels Reverse Transkriptease gewonnen wurde,
- der Anzahl von Zyklen die man nun fährt, um das Material mittels Polymerase zu vervielfältigen.
Die Amplifikation der cDNA — auch Vermehrung, Polymerisation oder Elongation genannt — wird also in Zyklen wiederholt. Nach einer bestimmten Anzahl von Zyklen — angezeigt als threshold cycle oder kurz Ct-Wert (40) — kann über geeignete Sonden (41) der Nachweis der gesuchten Gensequenz erbracht werden, weil diese nun in ausreichender Menge vorhanden ist. Das heißt nicht, dass beliebig viele Zyklen gefahren werden, bis man irgendein Positivergebnis findet — warum?
Wenn es um die angebliche Verbreitung des Coronavirus geht, ist regelmäßig von exponentieller Ausbreitung die Rede. Bewiesen wurde das nie. Konnte und kann es auch nicht, da ja bis zum heutigen Tage keine Ergebnisse von Baseline-Studien vorliegen, welche genau das liefern würden.
Bei der Polymerase hingegen ist die exponentielle Vermehrung real und gewollt, und da der Exponent den Wert zwei hat — was einer Verdoppelung im Intervall gleichkommt —, bedeutet dies, das in wenigen Dutzend Zyklen gigantische Quoten von amplifizierter cDNA erreicht werden können. Wie lässt sich das besser verbildlichen? Stellen wir uns dafür das folgende vor:
Nehmen wir an, dass im Abstrich eines Menschen zehn RNA-Sequenzen des E2-Gens vorhanden sind (was nichts darüber aussagt, ob da auch zehn “neuartige Coronaviren” sind). Das ist verschwindend wenig und mit Sicherheit völlig irrelevant für den betroffenen Organismus. Stellen wir uns im weiteren vor, dass von diesen zehn Minisequenzen eine die Tortur der Extraktion — der angeblichen Reinigung der Probe — überstanden hat. Vielleicht hat es sich aber auch im Material zufällig, zum Beispiel durch Mutationen gebildet, wer weiß das schon. Gehen wir jetzt davon aus, dass diese RNA mittels Reverse Transkriptase (RT) an DNS andocken konnte und die gewonnene cDNA auf ihre Vervielfältigung wartet. Dann sind wir nach einem Zyklus bei zwei, nach zwei Zyklen bei vier und nach zehn Zyklen bei 1024 durch Amplifikation gewonnenen Sequenzen, die uns das E2-Gen sichtbar machen könnten.
Das ist noch überschaubar. Aber wie sieht es nach 24 Zyklen aus? Dann sind wir bereits bei über 16 Millionen Treffern (16.777.216). Dieser Wert wurde aus einem einzigen Exemplar erfolgreicher RT gewonnen und hier wird auch klar, warum nach dieser Anzahl von Zyklen eine Weiterführung nicht mehr sinnvoll ist. Weil nun aus möglicherweise zufällig angefallenen, winzigen Genfragmenten gigantische Zahlen produziert werden können. Eine Aussage, welche die Kontamination der Probe im Hinblick auf das Vorhandensein oder gar die Menge des gesuchten Materials anzeigt, kann überhaupt nicht mehr getroffen werden. Somit kann es kaum überraschen, wenn Wissenschaftler davon abraten, die Amplifikation bei PCR-Tests, über eine Anzahl von 24 Zyklen hinaus weiterzuführen (42).
Und nun raten Sie mal, mit wie vielen Zyklen der Drosten-Test fährt, dazu noch einmal ein Zitat von weiter oben:
“Thermal cycling was performed at 55 °C for 10 min for reverse transcription, followed by 95 °C for 3 min and then 45 cycles of 95 °C for 15 s, 58 °C for 30 s.” (19iv)
Der Test aus der Charite, kreiert vom kritiklos gefeierten Team um Christian Drosten, definiert zum Nachweis von winzigen Genfragmenten, die man dem Genom des Coronavirus zuordnet, sage und schreibe 45 Zyklen.
Nach oben genanntem Beispiel kämen wir nach 45 Zyklen auf diesen Wert: 35.184.372.088.832. Ein Wert von 35 Billionen, gewonnen aus fast Nichts, ja möglicherweise aus einer oder mehreren Mutationen. Ein Wert der einen Test positiv kennzeichnet, obwohl in der Probe de facto NICHTS enthalten war. Es ist die Krone der Unwissenschaftlichkeit und der endgültige Beweis des vorsätzlichen Missbrauchs des Testes, um “laborbestätigte Fallzahlen” zu generieren.
Es kann für den Autor keine Zweifel geben, dass der Virologe Christian Drosten auch um das Problem einer zu hohen Anzahl gefahrener Zyklen und der Wertlosigkeit von daraus gewonnenen Ergebnissen weiß. Wer sonst sollte es besser wissen? Neben der WHO wissen es die Labore und erst recht weiß es Drosten (43, 44). Und trotzdem berät er die Bundesregierung auf der Basis von Zahlen, die mit einem gröblichst missbrauchten Test ermittelt wurden und zur Bewertung einer epidemiologischen Lage nie und nimmer herangezogen werden dürften. Er tut es wider besseren Wissens. Warum auch immer: Christian Drosten betrügt mit “seinem” Test Politiker, Behörden, Medien und die Bevölkerung. Und die wiederum lassen sich betrügen, weil sie aus diversen Gründen unfähig sind, diesen doch recht simplen Taschenspielertrick zu erkennen und vor allem anzuerkennen.
Die manipulative Anwendung des PCR-Tests durch das Fahren übermäßig vieler Zyklen zur Amplifizierung ist der offensichtlichste Betrug in einer ganzen Reihe weiterer. Würden die wohlbekannten Limits zur maximalen Anzahl von Zyklen eingehalten, wäre die angebliche Pandemie von einem Tag auf den anderen beendet. Und das unabhängig des Sachverhalts, dass da nicht einmal ein Virus, sondern nur eine wirklich, winzig, winzig kleine Gensequenz nachgewiesen wird.
“Eine neue Studie der Infectious Diseases Society of America ergab, dass bereits bei 25 Zyklen 70% der “positiven” PCR-Tests gar keine “Fälle” sind, da das Virus nicht kultiviert werden könne. Bei einem CT von 35 ergab sich bei 97% der Testpositiven kein klinisches Bild.” (45, 46)
Auf einen Schlag läge die Anzahl täglicher “laborbestätigter Fälle” in Deutschland nicht mehr bei 10- bis 30.000 sondern zwischen 300 und 900. Entsprechend schrumpfte die Gesamtzahl der in Deutschland “an oder mit Covid-19 Gestorbenen” auf etwa 1.500, was nicht einmal mit einer durchschnittlichen Grippewelle konkurrieren könnte. PCR-Tests die über 35 Zyklen hinaus amplifizieren, liefern schlicht Datenschrott (47) und gleichzeitig Propagandamaterial, um eine Pandemie, die keine ist, weiter zu befeuern.
Obiges ließe dabei immer noch außer acht, dass nur auf Minisequenzen von Genen getestet, Spezifitätstests mangelhaft oder gar nicht durchgeführt werden, die Extraktion der “Isolate” diskussionswürdig ist, verbindliche Standards für den Test nicht existieren, die Probenentnahme keinerlei Ansprüchen auf Repräsentativität und Transparenz genügt, Mehrfachtestungen gang und gäbe sind und diese “laborbestätigten Fälle” eine Untersuchung auf Symptomatiken nicht beinhalten. Allein der Umgang mit der Zyklenzahl zur Amplifizierung der Erbsubstanz weißt uns darauf hin, dass:
diese Pandemie erstunken und erlogen ist!
Wir können davon ausgehen, dass es genug Labore gibt, welche diese ja im “Goldstandard” vorgeschriebenen 45 oder zumindest mit weit über 30 Zyklen fahren (48, 49). Wie die Positivergebnisse auf SARS-CoV-2 zustande kamen, müssen die Labore auch nicht offen kommunizieren (50, 51). Es gibt diesbezüglich keine verbindlichen Standards (a8). Ein höchst erstaunliches Gebaren, wo es doch angeblich um Leben und Tod geht und deshalb Transparenz oberstes Gebot wäre.
Keine Suche nach Viren!
Selbst wenn die seit fast einem Jahr betriebenen Tests in der Lage wären, ein “neuartiges Virus” in seiner Gänze nachzuweisen, so genügten diese trotzdem in keiner Weise, um Infektionen oder gar Krankheiten nachzuweisen. Dieser Sachverhalt gilt unabhängig davon, ob es das SARS-CoV-2 — Virus nun gibt oder nicht. Niemals ersetzen Tests Diagnosen und erst recht stellen Tests keine Krankheiten fest!
Krankheiten äußern sich durch Symptome und die äußern sich zuerst in der subjektiven Befindlichkeit des Patienten. Auf welche Krankheit diese Symptome hinweisen könnten, kann ein Arzt feststellen, der nicht nur den Patienten untersucht, sondern sich auch mit dessen diesbezüglich relevanten Biografie bekannt macht (Anamnese). Diagnosen sind immer individuell und nicht automatisierbar.
Was jedoch immer und immer wieder unter den Tisch gekehrt wird, ist der Fakt, dass die PCR-Methode niemals nach Viren, sondern nach Gensequenzen, nach RNS (RNA) oder DNS (DNA) sucht. Diese Gensequenzen sind dazu noch extrem kurz und sie als solche exklusiv einem Erreger zuzuschreiben, hat mit Wissenschaft überhaupt nichts, mit Ideologie und der Durchsetzung politischer Agenden um so mehr zu tun.
Das ist auch der Grund, warum die Hersteller von PCR-Testkits zwar seit Monaten glänzende Geschäfte machen, sich aber aus der Haftung wohl bedacht herausnehmen. Sie weisen explizit darauf hin, dass der PCR-Test nicht zur Diagnose geeignet ist. So lesen wir zum Beispiel bei Creative Diagnostic:
“Dieses Produkt dient zum Nachweis für das 2019-Novel Coronavirus (2019-nCoV). Der Nachweis auf Basis dieses Produkts dient lediglich als klinische Referenz und sollte nicht als alleiniger Nachweis für die klinische Diagnose und Behandlung verwendet werden.” (52)
Der Pharmariese Merck warnt mehrfach sowohl bei der Nutzung seiner beworbenen Sonden und Primer für SARS-CoV-2 – Gensequenzen, als auch bei der Auswertung der Tests:
“Nur für Forschungszwecke. Nicht zur Verwendung in diagnostischen Verfahren vorgesehen.” (53)
Der US-amerikanische Anbieter BIOMEME erinnert:
“Positive Ergebnisse sind ein Hinweis auf das Vorhandensein von SARS-CoV-2 — RNA. Ein klinischer Abgleich mit Patientenanamnese und anderen diagnostischen Verfahren ist erforderlich, um den Infektionsstatus des Patienten zu bestimmen. Positive Ergebnisse schließen eine bakterielle Infektion oder eine Co-Infektion mit anderen Viren nicht aus. Der nachgewiesene Erreger [nachgewiesen ist die RNA-Sequenz] ist möglicherweise nicht die eindeutige Ursache der Erkrankung” (54)
Auf diese Weise sichern sich alle Hersteller ab, sowohl die von Testkits als auch die von Anlagen zur labortechnischen Auswertung (55, 56).
Das schert weder den RKI-Chef Lothar Wieler noch die Bundes- und Landesbehörden, erst recht nicht die Massenmedien und führende politische Sprachrohre. Es schert auch nicht angebliche Gesundheitsexperten wie den Hysteriker Karl Lauterbach und auch nicht gefeierte Virologen wie Christian Drosten. Der PCR-Test und die Diagnose von Covid-19 ist bei diesen Leuten das Gleiche. Und diese Leute wissen, dass sie lügen.
Daher lässt sich mit Fug und Recht behaupten:
Der PCR-Test kann keine SARS-CoV-2 – Viren identifizieren, sein technologischer Ansatz lässt es nicht zu. Damit kann er auch niemals Aussagen zu Infektionen oder gar Krankheiten treffen, sondern allenfalls Indizien liefern — und mehr nicht. In Bezug auf das “neuartige Virus” testet er auf extrem kurze, dem Virus zugeschriebene Gensequenzen. Wer von Covid-19 – Fällen in Folge positiv ausgefallener PCR-Tests spricht, verbreitet Unwahrheiten.
Das hat weitere Konsequenzen:
Eine Verbindung von sogenannten laborbestätigten Fällen auf Positivergebnisse von RNA-Sequenzen zum Einen und Krankheiten und Todesfällen zum Anderen herzustellen, ist völlig willkürlich und in keiner Weise kausal.
Außerdem: Der wissenschaftliche Beweis, dass die durch den PCR-Test angezeigten Gensequenzen ursächlich verantwortlich für die Covid-19 zugeschriebenen Krankheitssymptome wären, wurde niemals erbracht!
Daher sind alle Maßnahmen im Rahmen der sogenannten nichtpharmazeutischen Intervention nicht nur unverhältnismäßig und rechtswidrig. Sie sind kriminell. Denn als Basis dient ein Betrugsmodell und dafür missbraucht man bis zum heutigen Tage den PCR-Test. Kriminelles Handeln sollten wir nicht unterstützen, es dafür aber — wo immer es uns möglich ist — aufdecken. Wir sollten uns auch nicht weiter kriminellem Handeln unterwerfen. Die Zeit ist reif.
Bitte bleiben Sie sehr aufmerksam, liebe Leser.
Anmerkungen und Quellen
(Allgemein) Dieser Artikel von Peds Ansichten ist unter einer Creative Commons-Lizenz (Namensnennung – Nicht kommerziell – Keine Bearbeitungen 4.0 International) lizenziert. Unter Einhaltung der Lizenzbedingungen kann er gern weiterverbreitet und vervielfältigt werden. Bei Verlinkungen auf weitere Artikel von Peds Ansichten finden Sie dort auch die externen Quellen, mit denen die Aussagen im aktuellen Text belegt werden.
(a1) So es der Autor richtig versteht, haben die Isolate von Patientenproben, welche man kultiviert, um DNS- oder RNS-Sequenzen zu detektieren, mitnichten etwas mit einem (isolierten) Virus zu tun.
(a2) Da wir uns im deutschen Sprachraum bewegen, benutzt der Autor auch die deutschsprachigen Ausdrücke RNS (für Ribonukleinsäure) und DNS (Desoxyribonukleinsäure). In den Medien finden wir in der Mehrzahl die englischsprachigen Pendants RNA (RiboNucleid Acid) und DNA (DeoxyriboNucleid Acid); siehe: Ribonukleinsäure; https://de.wikipedia.org/wiki/Ribonukleins%C3%A4ure; und: Desoxyribonukleinsäure; https://de.wikipedia.org/wiki/Desoxyribonukleins%C3%A4ure; jeweils abgerufen: 22.01.2021
(a2) Übersetzungen aus dem Englischen unter Zuhilfenahme von DeepL.org
(a3) Jeden Hinweis auf eine möglicherweise ja doch existierende wissenschaftlich begründete Dokumentation zum vollständigen Nachweis des “neuartigen Virus” nehme ich gern entgegen und werde diesen gründlich prüfen. Hierzu werde ich den Rat von Medizinern und Mikrobiologen hinzuziehen.
(a4) Die PCR-Methode zielte ursprünglich auf die Kontrolle von Rein- und Reinsträumen zur Herstellung von Mikro-Chips. Sie wurde in den 1980er Jahren im Zuge der AIDS-Forschung für die Medizin entdeckt und sollte — mit sehr, sehr hoher Sensitivität designed — sicher die RNS des HI-Virus nachweisen.
(a5) RT-qPCR steht für Reverse Transcription Quick Time Polymerase Chain Reaction, das kleine ‘q’ (Quick Time) weißt darauf hin, dass der Test in Echtzeit Positivwerte signalisiert.
(a6) Das E-Gen als Ganzes ist 228 Basen lang. Der PCR-Test detektiert nur auf einen Teil dieses Gens, genannt E2, welches 76 Basen lang ist; siehe: 18.03.2020; NCBI, Gendatenbank; Severe acute respiratory syndrome coronavirus 2 isolate Wuhan-Hu-1, complete genome; https://www.ncbi.nlm.nih.gov/nuccore/MN908947.3?from=26245&to=26472
(a7) Zum Vergleich vom US-Gesundheitsministerium vorgegebene Primer-Konzentrationen, man betrachte die Werte für das N-Gen: 29.05.2020; Center for Disease Control and Prevention (CDC); 2019-Novel Coronavirus (2019-nCoV) Real-time rRT-PCR Panel — Primers and Probes; https://www.cdc.gov/coronavirus/2019-ncov/downloads/rt-pcr-panel-primer-probes.pdf; “ZEN™ Internal Quencher positioned between the ninth (9th) and tenth (10th) nucleotide base in the oligonucleotide sequence and Iowa Black® FQ (3IABkFQ) located at the 3’-end (Integrated DNA Technologies, Coralville, IA).”
(a8) Über vertrauenswürdige Quellen erfuhr ich, dass in Deutschland zwar auch Labore gibt, welche Tests mit vernünftigen Ct-Werten fahren, aber insgesamt mit den Empfehlungen des Drosten’schen “Goldstandard” hantiert wird, Zitat aus Quelle des Autor: “Auf telefonische Nachfrage hin erklärte man kürzlich, mehr als 30 Zyklen würde konkret 45 Zyklen heißen. Von anderen Laboren weiß ich, daß die Zyklenzahl auf 27 begrenzt wird.”
(1) 29.01.2020; The Lancet; Roujian Lu, Xiang Zhao, Juan Li und weitere; Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding; Lancet 2020; 395: 565–74; Published Online January 29, 2020; https://doi.org/10.1016/S0140-6736(20)30251-8; https://www.thelancet.com/pdfs/journals/lancet/PIIS0140-6736(20)30251-8.pdf
(2) 16.01.2020; Charité; Erster Test für das neuartige Coronavirus in China entwickelt; https://www.charite.de/service/pressemitteilung/artikel/detail/erster_test_fuer_das_neuartige_coronavirus_in_china_entwickelt/
(3) 20.02.2020; Na Zhu, Ph.D., Dingyu Zhang, M.D., Wenling Wang, und weitere; A Novel Coronavirus from Patients with Pneumonia in China, 2019; Kap. Isolation of Virus; https://www.nejm.org/doi/full/10.1056/nejmoa2001017
(4) 09.04.2013; Simply Science; Was ist DNA?; https://www.simplyscience.ch/teens-liesnach-archiv/articles/was-ist-dna.html
(5) 2020; Bayerisches Landesamt für Umwelt (LFU Bayern); DNA-Amplifikation (PCR); https://www.lfu.bayern.de/analytik_stoffe/biol_analytik_dna_untersuchungen/dna_amplifikation/index.htm
(6) 04.09.2018; DocCheck Flexikon; Patrick Messner, Frank Antwerpes, Norbert Ostendorf; Sanger-Sequenzierung; https://flexikon.doccheck.com/de/Sanger-Sequenzierung
(7) Oktober 2020; Corona Fakten; Samuel Eckert; Die Irrtümer der Virologen und die Binder-Labs; https://samueleckert.net/die-irrtuemer-der-virologen-und-die-binder-labs/
(8) 18.12.2018; BMC Informatics; Joshua B. Singer, Emma C. Thomson, John McLauchlan und weitere; GLUE: a flexible software system for virus sequence data; https://bmcbioinformatics.biomedcentral.com/articles/10.1186/s12859-018-2459-9
(9) https://en.wikipedia.org/wiki/List_of_sequence_alignment_software; abgerufen: 15.12.2020
(10) Nakamura T, Yamada KD, Tomii K, Katoh K. Parallelization of MAFFT for large-scale multiple sequence alignments. Bioinformatics 2018; 34: 2490–92.; verlinkt bei (1), S. 568
(11) Study Help; DNA / DNS Grundlagen; https://www.studyhelp.de/online-lernen/biologie/dna/; abgerufen: 13.12.2020
(12) 15.05.2003; nature; Koch’s postulates fulfilled for SARS virus; https://www.nature.com/articles/423240a; Fouchier, R., Kuiken, T., Schutten, M. et al. Koch’s postulates fulfilled for SARS virus. Nature 423, 240 (2003). https://doi.org/10.1038/423240a
(13) 03.07.2020; Corona-Fakten; Führende Corona Forscher geben zu, dass sie keinen wissenschaftlichen Beweis für die Existenz eines Virus haben; https://telegra.ph/Alle-f%C3%BChrenden-Wissenschaftler-best%C3%A4tigen-COVID-19-existiert-nicht-07-03
(14) 24.01.2020; The Lancet; Chaolin Huang, Yeming Wang, Xingwang Li und weitere; Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China; https://www.thelancet.com/journals/lancet/article/PIIS0140-6736(20)30183-5/fulltext
(15, 15i) Prinzip der Polymerase-Kettenreaktion (PCR); https://de.wikipedia.org/wiki/Reverse-Transkriptase-Polymerase-Kettenreaktion#Prinzip; abgerufen: 14.01.2021
(16) 2013; Kai Ilmo Ehnts; Entwicklung von Rekombinase-Polymerase-Amplifikations-Nachweisverfahren für virale Erreger von Atemwegsinfektionen; https://ediss.uni-goettingen.de/bitstream/handle/11858/00-1735-0000-0001-BAD4-F/Dissertation%20Ehnts.pdf?sequence=1; S. 27, Kap. Methoden; Anmerkung des Autors: Eine gut verständliche, straff geführte Abhandlung zum Nachweis von Influenza-Viren zugeschriebenen Gensequenzen
(17) Wissenschaft Plus; Stefan Lanka; Virologen die krankmachende Viren behaupten sind Wissenschaftsbetrüger und strafrechtlich zu verfolgen; http://wissenschafftplus.de/uploads/article/wissenschafftplus-virologen.pdf; S. 6
(18) 16.06.2020; EMM; Myungsun Park, Joungha Won, Byung Yoon Choi & C. Justin Lee; Optimization of primer sets and detection protocols for SARS-CoV-2 of coronavirus disease 2019 (COVID-19) using PCR and real-time PCR; Kap. RNA extraction; Kap. Materials and methods; https://www.nature.com/articles/s12276-020-0452-7
(19 bis 19iv) 23.01.2020; Eurosurveillance; Victor M. Corman, Olfert Landt, Marco Kaiser, Christian Drosten und weitere; Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR; Kap. Real-time reverse-transcription PCR; https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6988269/
(20) 20.02.2020; NEJM; Na Zhu, Dingyu Zhang, Wenling Wang und weitere; A Novel Coronavirus from Patients with Pneumonia in China, 2019; https://www.nejm.org/doi/full/10.1056/nejmoa2001017
(21) Sanger-Sequenzierung innerhalb der PCR; https://de.wikipedia.org/wiki/Frederick_Sanger#Methode_zur_Sequenzierung_von_Nukleins%C3%A4uren; abgerufen: 22.01.2021
(22) 14.01.2020; NCBI; GenBank: MN908947.2; Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome; https://www.ncbi.nlm.nih.gov/nuccore/MN908947.2
(23) 18.03.2020; NCBI; Severe acute respiratory syndrome coronavirus 2 isolate Wuhan-Hu-1, complete genome; https://www.ncbi.nlm.nih.gov/nuccore/MN908947
(24) Eurofins Genomics; rRT-PCR Primers & Probes for nCoV Coronavirus, Primers and probes recommended by the CDC & WHO; https://eurofinsgenomics.eu/en/dna-rna-oligonucleotides/optimised-application-oligos/ncov-qpcr-assays/; abgerufen: 17.12.2020
(25) 20.03.2020; AGES — Wissen aktuell; Vergleich unterschiedlicher Nukleinsäure-Amplifikationssysteme für SARS-CoV-2; https://www.ages.at/wissen-aktuell/publikationen/vergleich-unterschiedlicher-nukleinsaeure-amplifikationssysteme-fuer-sars-cov-2/
(26) WHO; Diagnostic detection of Wuhan coronavirus 2019 by real-time RT-PCR-Protocol and preliminary evaluationas of Jan 13, 2020; Victor Corman, Tobias Bleicker, Sebastian Brünink, Christian Drosten — Charité Virology, Berlin, Germany; Olfert Landt, Tib-Molbiol, Berlin, Germany; https://www.who.int/docs/default-source/coronaviruse/wuhan-virus-assay-v1991527e5122341d99287a1b17c111902.pdf
(27) cDNA oder komplementäre DNS; https://de.wikipedia.org/wiki/CDNA; abgerufen: 23.01.2021
(28) Biologie-Seite; Ribonuklease; https://www.biologie-seite.de/Biologie/Ribonuklease; und: Ribonukleasen; https://de.wikipedia.org/wiki/Ribonukleasen; jeweils abgerufen: 24.01.2021
(29) 16.01.2020; DZIF; Erster Test für das neuartige Coronavirus in China ist entwickelt; https://www.dzif.de/de/erster-test-fuer-das-neuartige-coronavirus-china-ist-entwickelt
(30, 30i) PCR-Optimierung, Degenerierte Primer, Forward und Reverse Primer-Design; https://de.wikipedia.org/wiki/Primerdesign#Degenerierte_Primer; abgerufen: 24.01.2021
(31) Molbiol; Designer für PCR-Primer; https://molbiol-tools.ca/PCR.htm; abgerufen 10.12.2020
(32) 27.11.2020; Pieter Borger, Bobby Rajesh Malhotra, Michael Yeadon; External peer review of the RTPCR test to detect SARS-CoV-2 reveals 10 major scientific flaws at the molecular and methodological level: consequences for false positive results.; B) Methods And Results, 1. Primer & Probe Design; 1a) Erroneous primer concentrations; https://cormandrostenreview.com/report/
(33) 30.11.2020; Antrag zur Zurückziehung wegen Mängeln; https://2020news.de/drosten-pcr-test-studie-rueckzugsantrag-gestellt-wegen-wissenschaftliche-fehler-und-massiver-interessenkonflikte/; siehe auch: 28.11.2020; https://cormandrostenreview.com/retraction-request-letter-to-eurosurveillance-editorial-board/
(34) 11.01.2021; Addendum zum Rückzugsantrag; https://2020news.de/20-gegen-drosten-neues-kapitel-im-retraction-prozess/; siehe auch: https://cormandrostenreview.com/addendum/
(35) 06.12.2020; Fassadenkratzer; Internationale Experten: Drosten-PCR-Test wegen schwerwiegender Mängel völlig untauglich für Infektions-Nachweis; https://fassadenkratzer.wordpress.com/2020/12/06/internationale-experten-drosten-pcr-test-wegen-schwerwiegender-mangel-vollig-untauglich-fur-infektions-nachweis/
(36) 01.06.2020; Theranostics; Dandan Li, Jiawei Zhang, Jinming Li; Primer design for quantitative real-time PCR for the emerging Coronavirus SARS-CoV-2; Table 2; https://www.thno.org/v10p7150.htm
(37) Ablauf der Polymerase-Kettenreaktion; https://de.wikipedia.org/wiki/Polymerase-Kettenreaktion#Ablauf; abgerufen: 23.01.2020
(38) Synthese von cDNA; https://de.wikipedia.org/wiki/CDNA#Synthese; abgerufen: 23.01.2021
(39) Bayerisches Landesamt für Umwelt; DNA-Amplifikation (PCR); https://www.lfu.bayern.de/analytik_stoffe/biol_analytik_dna_untersuchungen/dna_amplifikation/index.htm; abgerufen: 23.01.2021
(40) 15.07.2020; Bioscientia; Was bedeuten die Begriffe Dual-Target-PCR und Ct-Wert?; https://www.bioscientia.de/de/home/aktuelles/2020/07/was-bedeuten-die-begriffe-dual-target-pcr-und-ct-wert
(41) 02.06.2020; Medica Magazin, Universität Konstanz; Covid-19-Schnelltestverfahren auf Basis von DNA-Polymerasen; https://www.medica.de/de/News/Archiv/Covid-19-Schnelltestverfahren_auf_Basis_von_DNA-Polymerasen
(42) 10.06.2020; DGN; Jared Bullard, Kerry Dust, Duane Funk und weitere; Vorhersage der Infektiosität von SARS-CoV-2 bei positiver PCR; https://dgn.org/neuronews/journal_club/vorhersage-der-infektiositaet-von-sars-cov-2-bei-positiver-pcr/; übersetzt aus: 22.05.2020; Oxford Academic; Predicting Infectious Severe Acute Respiratory Syndrome Coronavirus 2 From Diagnostic Samples; https://academic.oup.com/cid/article/71/10/2663/5842165; auch bei PubMed; Predicting infectious SARS-CoV-2 from diagnostic samples; https://pubmed.ncbi.nlm.nih.gov/32442256/
(43) 05.08.2020; MVZ Labor Ravensburg; Was bedeutet der Ct-Wert?; https://www.labor-gaertner.de/fileadmin/user_upload/Dokumente/pdf/Aktuelles/LaborwissenKonrekt_SARS-CoV-2_Ct-Wert.pdf
(44) 2014; Wirtschaftswoche; Christian Drosten im Gespräch; “Der Körper wird ständig von Viren angegriffen”; https://www.wiwo.de/technologie/forschung/virologe-drosten-im-gespraech-2014-die-who-kann-nur-empfehlungen-aussprechen/9903228-2.html
(45) 28.09.2020; Oxford Academic; Rita Jaafar, Sarah Aherfi, Nathalie Wurtz und weitere; Correlation Between 3790 Quantitative Polymerase Chain Reaction–Positives Samples and Positive Cell Cultures, Including 1941 Severe Acute Respiratory Syndrome Coronavirus 2 Isolates; https://academic.oup.com/cid/advance-article/doi/10.1093/cid/ciaa1491/5912603
(46) 08.12.2020; Zeitpunkt; Erstmals verlangt eine Behörde die Offenlegung der Anzahl Zyklen beim PCR-Test; https://www.zeitpunkt.ch/erstmals-verlangt-eine-behoerde-die-offenlegung-der-anzahl-zyklen-beim-pcr-test
(47) 24.11.2020; Prof. Harald Wallach; Masken, Intensivbetten, PCR-Tests, Impfungen – Die apokalyptischen Reiter der Coronakrise stolpern durch den Wald der Fakten; Kapitel: Der PCR-Test und seine Aussagekraft; https://harald-walach.de/2020/11/24/masken-intensivbetten-pcr-tests-impfungen-die-apokalyptischen-reiter-der-coronakrise-stolpern-durch-den-wald-der-fakten/#more-2894
(48) 18.03.2020; AGES, Ingenetix; Vergleich unterschiedlicher Nukleinsäure-Amplifikationssysteme für SARS-CoV-2; Florian Gruber, Peter Hufnagel; https://www.ingenetix.com/wp-content/uploads/2020/03/Vergleichsstudie_SARS-CoV-2.pdf; S. 1
(49) 07.12.2020; Science Files; PCR-Tests: Viel (?) zu hohe Fallzahlen; https://sciencefiles.org/2020/12/07/pcr-tests-viel-zu-hohe-fallzahlen/; siehe auch Grafik im Text
(50) 07.10.2020; ARD-Tagesschau; Markus Grill, Mara Leurs; Gesundheitsämter bekommen keine Laborwerte; https://www.tagesschau.de/investigativ/ndr-wdr/gesundheitsaemter-corona-tests-103.html
(51) 10.10.2020; Bayerische Landesärztekammer; Pressemeldung: Aussagekraft von PCR-Tests auf SARS-CoV-2 erhöhen; https://www.blaek.de/meta/presse/presseinformationen/presseinformationen-2020/aussagekraft-von-pcr-tests-auf-sars-cov-2-erhoehen
(52) 21.03.2020; Creative Diagnostics; SARS-CoV-2 Coronavirus Multiplex RT-qPCR Kit; http://web.archive.org/web/20200321205248/https://www.creative-diagnostics.com/sars-cov-2-coronavirus-multiplex-rt-qpcr-kit-277854-457.htm
(53) Merck; Fallstudie zum Coronavirus-qPCR-Design & Produkte für die SARS-CoV-2-Forschung; https://www.sigmaaldrich.com/germany/ncov-coronavirus.html; abgerufen: 17.12.2020
(54) 20.08.2020; Food and Drug Administration USA (FDA); Biomeme; Biomeme SARS-CoV-2 Real-Time RT-PCR Test; https://www.fda.gov/media/141052/download; S.4/5
(55) Food and Drug Administration USA (FDA); Kogene Biotech; https://www.fda.gov/media/140069/download; S. 2; abgerufen: 17.12.2020
(56) 17.12.2020; Roche; Allgemeine Informationen zu SARS-CoV-2; https://www.roche.de/diagnostik-produkte/produktkatalog/tests-parameter/sars-cov-2-cobas-6800-8800/
(b1, b1i, b1ii) EMM; PCR primer design and optimization guidelines, SARS-CoV-2 genome map, and targets of primer sets; https://www.nature.com/articles/s12276-020-0452-7/figures/1; entnommen: 12.12.2020
(b2) Die Doppelhelix wird durch die Helikase und die Topoisomerase geöffnet. Danach setzt die Primase einen Primer und die DNA-Polymerase beginnt, den leading strand zu kopieren. Eine zweite DNA-Polymerase bindet den lagging strand, kann aber nicht kontinuierlich synthetisieren, sondern produziert einzelne Okazaki-Fragmente, welche von der DNA-Ligase zusammengefügt werden.; Autor: Madprime; September 2010; Quelle: Wikimedia; https://upload.wikimedia.org/wikipedia/commons/3/33/DNA_replication_split_horizontal.svg; Lizenz: GNU Free Documentation License
(b3) siehe (19)
(Titelbild) Mikroskop, Analyse, Labor; Autor: Chokniti Khongchum (Pixabay); 13.04.2019; https://pixabay.com/de/photos/analyse-becher-biochemie-biologie-4402809/; Lizenz: Pixabay License

